Введение
В первой части мы сравнили ДНК и клеточные механизмы с машинным кодом неизвестной программы – огромным бинарным «черным ящиком», который учёные столетиями пытаются реверс-инжинировать, не имея исходного кода. Мы рассматривали клетку как компьютер: ДНК – как двоичный код программы, органоиды – как модули процессора, а сама клетка – как вычислительная машина. Однако до конца понятно не что делает эта программа и как она работает, поэтому исследователям приходится действовать как хакерам-реверсерам – через анализ входов/выходов и постепенное вскрытие внутренних механизмов.
Теперь, во второй части, мы сосредоточимся на новом подходе: рассматривать отдельную клетку как виртуальный город, где множество компонентов взаимодействуют подобно жителям, зданиям и инфраструктуре города. Вместо детального моделирования каждого атома мы будем оперировать более крупными единицами – молекулами, белками, органеллами – примерно так же, как игровые движки оперируют объектами (машинами, персонажами, зданиями) вместо отслеживания движения каждого электрона. Такой уровень абстракции позволит нам изолировать отдельные процессы в клетке, взяв точку отсчёта – конкретный элемент или событие – и проследить его взаимодействия с окружением шаг за шагом.
Попробуем представить клетку «изнутри» – как если бы мы могли уменьшиться до микроскопических размеров и наблюдать за происходящим, подобно герою детектива или персонажу научно-фантастической игры от первого лица. Что это даёт науке? В этой статье мы порассуждаем, как структурный, многоуровневый подход с понятными аналогиями из ИТ способен улучшить и ускорить исследование живых клеток. Мы рассмотрим 10 конкретных способов, как “виртуальный город” из одной клетки и методы реверс-инжиниринга без исходного кода помогут быстрее понять работу клеточных механизмов. Каждый из этих способов мы подкрепим существующими примерами или экспериментальными данными и обозначим, чего ещё не хватает для реализации полного потенциала такого подхода.
(Примечание: Чтобы иллюстрировать идеи, мы будем использовать как концепт-арты, так и реальные научные изображения, доступные на сегодняшний день. Все ссылки и источники указаны в тексте.)
Клетка как виртуальный город
Представьте себе одиночную животную клетку как миниатюрный мегаполис. Вокруг – плазматическая мембрана, выполняющая роль городской стены или границы города, через которую проходят необходимые ресурсы и удаляются отходы. Внутри – целая инфраструктура: ядро выступает в роли городского архива или правительственного центра (именно там хранится генетический «код» – DNA – управляющий всеми процессами), митохондрии – электростанции, снабжающие город энергией, рибосомы – фабрики, производящие основные «товары» клетки – белки, эндоплазматический ретикулум и аппарат Гольджи – транспортные и логистические сети, распределяющие материалы, лизосомы – мусороперерабатывающие заводы, ликвидирующие отходы. Каркас города образует цитоскелет – сеть «дорог» и опор, по которым перемещаются грузы (везикулы) и поддерживается форма клетки.
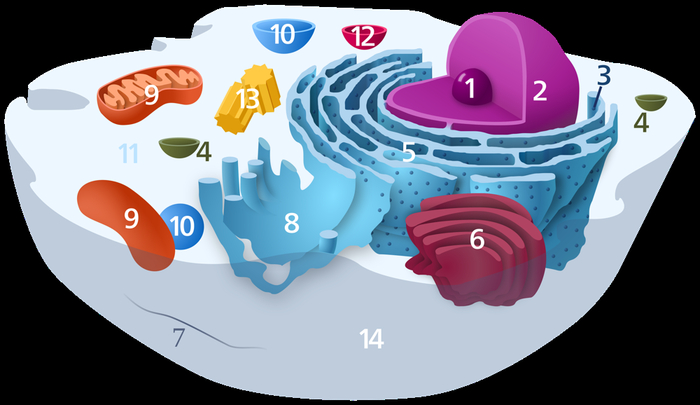
Схематическое строение типичной животной клетки (анагогично «городу»): 1 – ядро (центр управления), 2 – ядрышко (архив, где хранятся чертежи рибосом), 3 – рибосомы (фабрики белков), 4 – транспортные везикулы, 5 – шероховатый эндоплазматический ретикулум (конвейерная сеть сборки белков), 6 – аппарат Гольджи (логистический центр), 7 – цитоскелет (каркас и «улицы»), 8 – гладкий эндоплазматический ретикулум (химический завод по синтезу липидов), 9 – митохондрия (электростанция), 10 – вакуоль (склад, у животных клеток малый), 11 – цитозоль (внутренняя среда – «атмосфера» города), 12 – лизосома (переработка отходов), 13 – центриоли (организация «строительства» при делении), 14 – клеточная мембрана (граница города)[3][4].
Аналогия «клетка = город» не нова – её часто используют в образовании. Однако здесь она послужит не только наглядности, но и основой для моделирования: мы хотим буквально создать виртуальный прототип клетки. Подобно тому, как градостроительный симулятор моделирует жизнь города (с жителями, транспортом и экономикой), мы можем смоделировать клетку, где белки выступают как агенты-жители, потребляющие ресурсы и выполняющие работы, молекулы АТФ, аминокислоты, ионы – как товары и энергия, органеллы – как специализированные здания-фабрики. В таком виртуальном городе кипит жизнь: по «улицам» цитоплазмы перемещаются моторные белки, таская грузы, на митохондриальных «электростанциях» синтезируется энергия, в ядре принимаются «управленческие решения» в виде экспрессии генов.
Зачем нам такая картина? Дело в том, что простое перечисление деталей клетки мало помогает понять динамику её работы. Можно знать «список улиц и зданий» города, но не понимать, как происходит утренний час пик, куда бегут потоки людей, что произойдёт, если перекрыть мост или отключить электростанцию. Точно так же в клетке важно увидеть процессы во времени и пространстве: кто с кем взаимодействует, куда идут потоки веществ, что критично для выживания. В реальном эксперименте заглянуть внутрь клетки сложно – мы либо наблюдаем общую картину под микроскопом (и видим лишь крупные структуры), либо изучаем отдельные молекулы (теряя контекст целого). Виртуальная модель же может дать панорамный обзор всех событий одновременно, позволяя изолировать и изучать интересующие процессы.
Уровни абстракции в биологии
Чтобы осуществить такую модель, необходимо правильно выбрать уровень абстракции – аналогично тому, как в программировании и электронике используют многоуровневую архитектуру (битовый уровень, ассемблер, язык высокого уровня и т.д.). Биологические системы тоже можно разложить на иерархию уровней: атомы → молекулы → макромолекулы (белки, нуклеиновые кислоты) → надмолекулярные комплексы → органеллы → клетка → ткань и т.д. Очевидно, моделировать каждый атом во всей клетке (а их там порядка $10^{14}$) нереально – ни вычислительных мощностей, ни смысла в таком низком уровне для понимания общей картины нет. Намного продуктивнее подняться на несколько уровней выше, рассматривая, например, белки целиком как единицы (пусть и с учётом их основных свойств), а взаимодействия – как события (реакции, связывания), а не как столкновения отдельных атомов.
Такой подход соответствует принципам опытных реверс-инженеров: работать на максимально высоком уровне, не теряя сути проблемы. В программном анализе это значит думать как разработчик, а не следовать пословно машинному коду[5]. В биологии это значит оперировать понятиями функций и путей, а не тоннами сырых химических деталей. Новички часто тонут в деталях, пытаясь учесть каждую молекулу, каждый квант; эксперты же вводят абстракции – ключевые переменные и модули – и углубляются в детали лишь когда это необходимо для понимания проблемы[6].
Идея многоуровневой архитектуры клетки была предложена и в смежных областях, например, в синтетической биологии. Ещё в 2006 году исследователи прямо сравнили биологическую систему с компьютером: ДНК, РНК, белки и метаболиты – это физический уровень «железа» (аналог транзисторов и конденсаторов), биохимические реакции – уровень логических элементов (гейтов), контролирующих потоки информации, функциональные модули из реакций – подобие интегральных схем или подпрограмм, которые можно комбинировать[7]. Объединяя модули и подключая их к общей «шине» клетки, синтетический биолог может запрограммировать новую функцию клетки, как программист собирает систему из библиотек[8]. Нам же важна обратная задача – разобрать уже существующую клетку по уровням, чтобы понять, как каждый уровень рождает следующий.
Практически это значит, что при моделировании мы можем декомпозировать клетку на подсистемы: отдельно смоделировать, к примеру, генетическую регуляцию, сигнальные пути, метаболические сети, механическую работу цитоскелета и т.д., а затем связать эти подсистемы. Такой принцип уже применялся: в первой компьютерной модели полной клетки (бактерии Mycoplasma genitalium) Маркус Коверт с коллегами собрали 28 отдельных подмоделей разных процессов, от репликации ДНК до метаболизма[9], и запустили их в едином симуляторе, который шаг за шагом воспроизводил жизненный цикл бактерии[10]. Это был поразительный пример, как многослойный подход (каждый процесс описывается на своей абстракции и временном масштабе) позволяет охватить всю клетку целиком.
Важно отметить: выбор уровня детализации – это всегда баланс между достоверностью и вычислительной сложностью. Современные 3D-визуализации клеток зачастую умышленно упрощают образ для ясности: убирают «лишние» молекулы, снижают плотность среды, замедляют слишком быстрые движения – иначе зрителю будет ничего не разобрать[11][12]. Например, в проекте виртуальной реальности “Journey to the Centre of the Cell” авторам пришлось облегчить изображение клеточной поверхности, чтобы виртуальный тур работал с нужной частотой кадров[13]. Последовавший проект Nanoscape пошёл дальше: он интегрировал колоссальный объём научных данных в интерактивную сцену на игровом движке Unity3D, но тоже умеренно упростил некоторые аспекты (водную среду, мельчайшие молекулы), чтобы пользователь мог свободно перемещаться и наблюдать процессы[14][15]. Эти упрощения не искажают ключевых взаимодействий, но значительно снижают «шум» и нагрузку на систему. Так что, как ни парадоксально звучит, убрать лишнее – значит увидеть главное.
Изоляция процессов: взгляд изнутри клетки
Когда структура клеточного «города» понятна и уровни выбраны, можно переходить к изучению отдельных процессов в изоляции. В методологии реверс-инжиниринга программ есть принцип: не пытаться сразу понять весь код целиком, а выделить участок, отвечающий за интересующую функцию, и сосредоточиться на нём[16][6]. Аналогично в клетке мы можем выбирать конкретную точку отсчёта – скажем, молекулу, сигнал или событие – и прослеживать, как от неё расходятся волны взаимодействий.
Представьте, что мы «запускаем» виртуальную клетку и останавливаем время в определённый момент, как если бы поставили игру на паузу. Затем выбираем, к примеру, одну молекулу сигнального гормона, которая только что проникла через мембрану. Дальше шаг за шагом (или покадрово) наблюдаем, что происходит: гормон связывается с рецептором на мембране, рецептор меняет форму (наш виртуальный мир это отобразит упрощённо, без квантов химии, но с правильным исходом), затем рецептор активирует каскад белков-посредников в цитоплазме, сигнал добирается до ядра, где запускается экспрессия гена, и т.д. Мы как бы следуем конкретному маршруту по клеточному городу – например, от городских ворот (мембраны) до ратуши (ядра) через серию адресов – и отмечаем все задействованные элементы.
В реальном эксперименте учёные подобное делают косвенными методами – применяя меченые флуоресцентные молекулы, они могут видеть путь сигнала (цветные метки светятся по очереди, показывая активацию белков). Однако в один момент времени обычно отслеживают лишь несколько меток. В виртуальной же клетке мы можем отслеживать их десятки и сотни одновременно, получая полную картину. Это как в комплексном расследовании: вместо опроса по очереди отдельных свидетелей мы сразу видим всю сцену преступления целиком во всех деталях.
Кроме слежения за сигналом, изоляция процесса может означать проведение виртуального эксперимента над клеткой: например, выключение (нокаут) какого-то гена или блокировка работы органеллы – и наблюдение, как это повлияет на остальную систему. Биологи широко используют метод нокаута генов: убирают или подавляют один ген и смотрят, что сломается. Проблема в том, что в организме сотни генов действуют сообща, и эффект отключения одного может быть неочевиден или компенсирован другими путями[17]. Компьютерная модель клетки позволяет отключать сразу комбинации генов или параметров и видеть комплексный результат, делая то, что на живой клетке зачастую непрактично или слишком долго. Не зря Коверт и коллеги отмечали, что цель создания полной модели клетки – преодолеть разрыв между обилием данных и пониманием: интегрировать всё знание в одно место и «прокрутить» его во времени[18]. Так мы сможем найти неожиданные взаимосвязи, которые не видны при разрозненных экспериментальных данных.
Наконец, взгляд изнутри подразумевает, что исследователь становится как бы участником событий. Футуристично можно представить себе, что наука движется к инструментам, где учёный в VR-очках гуляет по виртуальной клетке, как герой фильма «Фантастическое путешествие», и собственноручно «трогает» молекулы, переставляет их или вручную запускает реакции, проверяя, что будет. От фантастики к реальности нас отделяют уже не такие уж непонятные задачи – в первую очередь, колоссальная работа по сбору и структурированию данных о всех элементах клетки, а также развитие интерфейсов для взаимодействия человека с такими огромными моделями. Многие части этой мозаики уже существуют в зачатке. Далее мы подробно рассмотрим 10 конкретных направлений, где подход виртуального города и реверс-инжиниринга клетки способен ускорить прогресс – и что уже достигнуто в каждом из них на сегодняшний день.
10 способов ускорить изучение работы клетки
1. Иммерсивная 3D-визуализация процессов в клетке
Виртуальный «тур» по клетке. Современные технологии позволяют создать научно достоверную трёхмерную сцену внутренней поверхности клетки, где исследователь может перемещаться от первого лица. На изображении показаны кадры из Nanoscape – интерактивной модели раковой клетки: на «поверхности» клетки видны рецепторы (белковые «антенны»), рядом изображены соседние клетки, волокна коллагена внеклеточного матрикса (зеленоватые нити) и капилляр с эритроцитами[19][20]. Пользователь словно уменьшен до 40 нанометров роста и гуляет по ландшафту, наблюдая происходящие процессы в реальном времени[14].
Подобная иммерсивная 3D-визуализация даёт возможность “увидеть невидимое”. Если традиционная микрофотография или схема предоставляют статическую и плоскую информацию, то в виртуальной клетке можно разглядывать структуры под любым углом и в динамике. Например, вместо абстрактных стрелочек на схеме сигналинга мы увидим, как молекулы реально диффундируют в цитоплазме, находят друг друга и взаимодействуют. Это помогает построить интуитивное понимание связей: учёный мгновенно замечает, что, допустим, митохондрии сгруппированы вокруг ядра, или что определённые белки сконцентрированы вблизи определённых структур. Там, где текст или 2D-график требует долгого анализа, визуальная сцена даёт целостный образ.
Уже показано, что такие визуализации заметно улучшают понимание сложных концепций даже у специалистов. В эксперименте с VR-проектом “Journey to the Centre of the Cell” студенты-биологи после виртуального тура по клетке намного лучше усвоили расположение и функции органелл[21]. Nanoscape, о котором упоминалось выше, изначально создавался как образовательный проект, но его авторы прямо задаются вопросом: а не поможет ли это самим исследователям лучше понять свои же данные?[22] Пока ещё трудно оценить, насколько 3D-модели дадут новые научные открытия, но есть большая надежда, что совмещение разных данных в одной пространственной модели натолкнёт на идеи, которые не возникают при разрозненном рассмотрении. К примеру, совмещение данных микроскопии, протеомики и транскриптомики в одной сцене может позволить визуально заметить корреляции – скажем, некоторые белки всегда рядом в пространстве и по времени синхронно действуют, что намекнёт на неизвестный комплекс или путь.
Добавим, что игровые движки и VR-интерфейсы делают работу с такой моделью более интерактивной: учёный может не только смотреть, но и “трогать” клетку, переключать слои информации, увеличивать или уменьшать масштаб (вплоть до отдельных молекул или наоборот – до уровня нескольких клеток). Например, в Nanoscape планируется реализовать возможность плавно менять масштаб детализации – от просмотра крупных процессов до отдельной атомной структуры – и настраивать «скрытие» части объектов[23]. Это сродни тому, как в Google Maps вы можете увидеть и план города, и отдельный дом при приближении. Для науки это означает более гибкий анализ: можно выявить общий эффект на уровне клетки, а затем мгновенно “приблизиться”, чтобы проследить механизмы на молекулярном уровне.
2. Абстрагирование: белки как объекты, а не наборы атомов
Одна из ключевых идей нашего подхода – упростить представление компонентов, сохранив их главное поведение. Ведь цель – понять функцию и взаимодействие элементов клетки, а не решать уравнения квантовой механики для каждой молекулы. Поэтому рассматриваем белок целиком как единицу (“агент”) с набором свойств (форма, заряды, сайты связывания), а не как тысячу отдельных атомов. Точно так же в современных симуляторах частиц часто моделируют целую пылинку как одну частицу, а не все молекулы в ней.
На практике такой подход уже используют в научной визуализации: например, в проекте Nanoscape для большей наглядности исключили молекулы воды и мелкие метаболиты (считается, что вода просто создаёт фон, не влияя на видимые процессы), вместо детальной модели липидного слоя мембраны применили анимированную текстуру, а движение белков умышлено замедлили, чтобы пользователь успел заметить их взаимодействия[11][24]. Результат – сглаженная “картинка”, где главное не теряется в мельчайшей дрожи и суете. Конечно, это визуальная абстракция, но аналогичные принципы работают и в математическом моделировании. К примеру, при расчётах метаболических потоков химические реакции описывают не квантово-механическими уравнениями, а более простыми кинетическими моделями или даже стехиометрическими балансами – то есть упрощают, предполагая, что детали столкновений несущественны, важно только сколько вещества превращается во сколько.
Белок как объект может обладать атрибутами: где находится, с чем может связаться, что случается при связи. Эти свойства можно взять из экспериментальных данных (структурные базы данных, кинетические параметры реакций и пр.). Тогда в симуляции белки будут «жить своей жизнью», взаимодействуя при случайных столкновениях в соответствии с заданными вероятностями, почти как в реальности – но без симуляции каждого шага молекулы воды вокруг них. Такой агент-ориентированный подход резко снижает вычислительную сложность, позволяя смоделировать больше и дольше. Вместо отслеживания $10^{14}$ атомов мы отслеживаем, скажем, $10^7$ молекул, что уже приближается к реализуемому на современных суперкомпьютерах.
Стоит подчеркнуть, что правильно выбранный уровень абстракции не только экономит ресурсы, но и проясняет логику системы. В программировании высокоуровневый код понятнее ассемблера, так и в клетке: наблюдать взаимодействия белков понятнее, чем смотреть на хаотические толчки атомов. Главное – убедиться, что наша абстракция не упустила критических эффектов. Например, в некоторых случаях физическая толпа молекул (эффект ограниченного диффузионного пространства) влияет на реакцию – тогда придётся включить параметр плотности среды или особые взаимодействия. Но такие эффекты тоже можно учесть статистически, не опускаясь до атомов.
В итоге, обращаясь к аналогии: мы рассматриваем город на уровне зданий и жителей, а не элементарных частиц материи. Нам важно, что два человека могут войти в контакт или нет, а из чего состоят атомы их тел – неважно для изучения транспортной системы или экономики города. Таким образом, мы очищаем модель от излишней информационной перегрузки и концентрируемся на функциональных отношениях.
3. Модульность и уровни – “архитектурный план” клетки
Город обычно делится на районы (промышленный, жилой, административный), и внутри каждого действуют свои правила и процессы. Также и клетку удобно разделить на функциональные модули: метаболический блок, генетический блок, блок сигналинга, и т.д. Это соответствует обсуждавшимся выше уровням абстракции и подсистемам. Такая структуризация ускоряет исследования сразу по нескольким причинам.
Во-первых, специализация: разные научные группы могут фокусироваться на разных модулях, а затем объединять результаты. Один коллектив подробно моделирует метаболизм (биохимики), другой – регуляцию генов (молекулярные биологи), третий – механику деления клетки (биофизики). Каждый работает со своей «частью города», используя наиболее подходящие методики, а потом через стандартизованные интерфейсы (условно – точки стыковки между путями) модули собираются вместе. Такой принцип давно предлагают в синтетической биологии: стандартизировать «биологические интерфейсы» наподобие электронной схемотехники[25][26]. Наш случай – обратный (мы не строим, а изучаем), но подход тот же – декомпозиция облегчает понимание.
Во-вторых, модульность позволяет изолированно тестировать части системы. Если есть подозрение, что сбой в клетке связан с метаболизмом – мы можем запустить симуляцию только метаболического модуля под разными условиями (например, варьируя наличие питательных веществ) и посмотреть, нет ли там узкого места. Аналогично, для генетической сети можно отдельно проверить, как колебания одного трансрипционного фактора влияют на остальные – без одновременного усложнения метаболизмом. Это сродни тому, как в софтверном проекте тестируют отдельные модули (unit testing) прежде, чем проверять всю систему целиком.
Иерархия уровней позволяет также связывать явления разного масштаба. Классический пример: мутация в ДНК (низший уровень) приводит к изменению структуры белка, что ломает работу какого-то органелл (средний уровень) и в итоге нарушает функцию клетки (верхний уровень). Используя многоуровневую модель, можно проследить эту цепочку: начать с изменения в «коде» (геноме) и увидеть, как оно отражается на поведении системы. В обратную сторону тоже работает – обнаружив странность в поведении клетки, можно по моделям “просверлить” вниз, выясняя, не является ли причина в изменении конкретного компонента.
Реальный прогресс в этом направлении демонстрирует уже упомянутый полный компьютерный прототип клетки Mycoplasma (2012 год): исследователи собрали по литературе информацию о каждой известной подсистеме клетки (всего 28 моделей) и сделали программу, шаг за шагом имитирующую жизнь бактерии[10][18]. Модель предсказывала в том числе время деления клетки, эффекты от нокаута генов и многие другие феномены. Это не была единая красивая 3D-картинка – скорее разнообразные алгоритмы, запущенные параллельно, – но сам факт, что целостная картина сложилась из модулей, вдохновил исследователей на дальнейшие проекты. Сейчас, спустя 10+ лет, идут работы над моделями более сложных клеток, включая модели минимальной синтетической клетки JCVI-syn3A (2021) – у которой около 500 генов. Там также применили модульный подход, но уже с более интегрированной, единой симуляцией всех процессов[27].
В итоге, чёткий структурный план клетки, как архитектурный чертёж, помогает понять “из чего она состоит” и как части взаимодействуют. Это создает язык описания, общий для разных экспертов, и упорядочивает накопленные данные. А как только есть порядок – появляются и новые возможности анализа, о которых мы поговорим дальше.
4. Виртуальные эксперименты: цифровой двойник клетки
Одно из главных преимуществ наличия цифровой модели клетки – возможность проводить на ней эксперименты, не трогая живые клетки. Такой in silico эксперимент может быть гораздо гибче и масштабнее, чем in vivo. Например, можно мгновенно “вырезать” любой компонент и тут же увидеть, к чему это приведёт – без сложных лабораторных процедур. Можно многократно повторять симуляцию, пробуя разные условия, и быстро собирать статистику.
Уже первые полные модели клетки показали мощь этого подхода. Созданная модель Mycoplasma genitalium позволила отвечать на вопросы, которые было крайне трудно или невозможно проверить на реальных бактериях[28]. Например, она помогла выяснить, какие гены наиболее важны для выживания клетки: исследователи виртуально “нокаутировали” по очереди все гены и смотрели, выживет модель или нет. Конечно, потом эти предсказания сравнили с настоящими бактериями. Более того, модель стала шагом к компьютерному биоинжинирингу – идее, что однажды можно проектировать и модифицировать живые клетки, как сейчас разрабатывают микросхемы[28].
Совсем свежий пример – проект минимальной синтетической клетки JCVI-syn3A, упомянутый выше. Учёные сначала синтезировали бактерию с минимальным набором генов (всего 493 гена, против ~4000 у стандартных бактерий) – это реальный организм, “поломанный” до базовых функций. Но 92 гена там всё ещё с неизвестной функцией[29]. Для такого упрощённого организма создали компьютерную модель, которая, будучи настроена, начала давать конкретные проверяемые прогнозы. Один из потрясающих результатов: модель предсказала, что если обратно добавить два гена (из ранее вырезанных, кодирующих ферменты пируватдегидрогеназы) в минимальную клетку, то клетка станет делиться быстрее – период деления сократится примерно на 13%[30]. Исследователи тут же проверили это в лаборатории: ввели эти два гена в реальную минимальную бактерию – и действительно, время между делениями уменьшилось с 120 до 105 минут (~12%)[31]. То есть цифровой двойник клетки фактически подсказал улучшение биологической функции, и подсказка оказалась верной почти точностыо! Это замечательный пример, как виртуальный эксперимент экономит массу времени: без модели пришлось бы гадать, какой из сотни удалённых генов стоит вернуть для ускорения роста, а тут выбор стал очевиден.
Виртуальные эксперименты помогают не только в поиске функций генов, но и, например, в разработке лекарств. Допустим, у нас есть модель раковой клетки – мы можем протестировать на ней тысячи потенциальных лекарственных молекул, просто включая их по одной и смотря, нарушают ли они какие-то процессы, важные для выживания клетки. Конечно, компьютерная модель – это лишь приближение, но она может отсеять заведомо неработающие варианты и сузить круг кандидатов, экономя ресурсы на реальных испытаниях. В будущем такая система может лечь в основу персонализированной медицины: имея цифровую модель конкретной клетки пациента (например, раковой клетки с её мутациями), можно пробовать разные схемы терапии на компьютере и выбирать наиболее эффективную комбинацию, прежде чем применять к человеку.
Важно понимать, что пока мы не в состоянии смоделировать абсолютно всё с идеальной точностью. Но по мере накопления данных и роста вычислительных мощностей цифровые двойники клеток становятся всё точнее. Мы уже видим, что компьютерные модели способны не только объяснять известное, но и предсказывать новое, что проверяется на практике[31]. А это значит, что реверс-инжиниринг клетки действительно начал работать: мы не просто беспомощно изучаем черный ящик, а строим его копию и экспериментируем на ней, как на открытой системе.